Site-directed mutagenesis
- Site-directed mutagenesis는 특정 DNA 염기서열을 변화시킴을 의미하는, 분자생물학에서 중요하게 이용되는 분야이다. 즉 어떤 단백질의 기능부위를 연구할 때에 의심되는 부위의 아미노산을 다른 아미노산으로 치환 혹은 제거한 다음 기능의 변화를 관찰함으로써 기능부위를 규명할 수 있다.
- 또 DNA 재결합시 필요한 제한효소 절단부위를 만들거나 소실시킬 때 사용되기도 한다.
- 또한 promoter나 enhancer 연구에서 어떤 transacting factor가 결합하여 기능을 하는지를 알기 위하여 그 결합 부위의 염기서열을 변화 시켜서 결합을 못하게 한 다음 promoter나 enhancer의 활성을 측정함으로써 그 transacting factor의 중요성을 타진할 수도 있다.
- 이상과 같이 site-directed mutagenesis는 여러 분야에 적용되는 강력한 도구가 되었으며, 여러가지 방법이 개발되어 소개되었다. 그 중에서 구체적인 실험 방법은 그중 간단하고 가장 많이 사용되고 있는 Site-directed mutagenesis (with PCR-Dpn I) 제시하고, 대표적인 3가지 방법만을 소개하고 자 한다.
Site-directed mutagenesis (with PCR-Dpn I)
- 실험 재료
- Pfu DNA polymerase(2.5 U/μl)
- 10x reaction buffer[100 mM KCl, 100 mM (NH4)2SO4, 20 mM Tris-Cl, pH 8.8, 20 mM MgSO4, 1% Triton X100, 1 mg/ml bovine serum albumin]
- Oligonucleotide primer: 원하는 mutation을 가지고 있는 서로 상보적인 두개의 primer가 필요하다. 보통 primer 크기는 30mer 내외로 하고 mutation 위치는 중앙에 위치하게 한다.
- dNTP mix(10 mM, each)
- Competent cell
- Dpn I(10 U/μl)
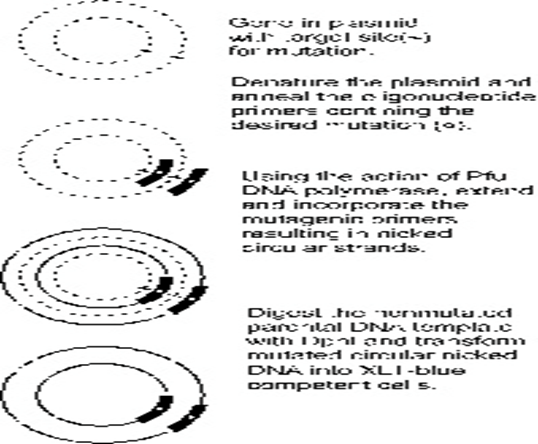
- 실험 방법
1. 아래와 같이 반응액을 PCR tube에 준비한다.
10x reaction buffer 5 μl
Plasmid DNA(10 ng/μl) 1 μl
Oligonucleotide primer #1(10 pmole/μl) 1.25 μl
Oligonucleotide primer #2(10 pmole/μl) 1.25 μl
dNTP mix 1 μl
ddH2O to 50 μl
2. 최종으로 Pfu DNA polymerase를 1 μl 첨가하고 섞은 후 PCR을 시행한다. PCR cycle 온도와 시간은 아래와 같이 한다.
95°C 30초, 1회
95°C 30초, 55°C 30초, 68°C 10분, 15회
4°C 보존
3. 반응이 종료되면 Dpn I(10 U/μl) 효소 1 μl를 직접 PCR 반응액에 첨가하고 섞은 후, 37°C에서 1 시간 반응시킨다.
4. Dpn I을 처리한 용액 1 μl로 XL1이나 DH5α 등의 E.coli에 transformation 시킨다.
첫번째 : Stratagene 회사에서 개발하여 판매하는 QuickChange Site-Directed Mutagenesis kit를 이용한 방법
(상기 소개된 실험 방법이다.)
- 모두 6시간 정도가 소요된다. Mutation을 가진 두개의 상보적인 염기서열의 oligonucleotide를 primer로 사용하고, plasmid 상태의 DNA를 template로 사용하여 새로이 mutation을 가진 plasmid를 시험관에서 증폭, 합성한 다음, 원래의 wild type은 Dpn I 제한효소로 절단하여 제거하는 방법이다.
- 보통 실험실에서 사용하는 E.coli는 Dam methylase를 가지고 있어서 이들에서 분리된 모든 DNA의 경우 GATC 염기서열의 A가 methylation 되어있다. 즉 template로 사용한 wild type DNA는 E.coli에서 분리된 DNA로서 Gm6ATC를 인식하여 절단하는 Dpn I에 의하여 절단되지만 시험관에서 합성한 mutant DNA는 절단되지 않는다.
- Dpn I으로 처리한 DNA를 E.coli host에 transformation 시킴으로써 높은 확률로 mutant를 가진 clone을 얻을 수 있다.
두번째 : uracil를 함유한 single-stranded DNA를 이용하는 방법
- 두개의 중요한 DNA repair 효소인 dUTPase(dut)와 uracil N-glycosylase(ung) 유전자의 결함이 있는 E.coli strain에서는 thymine 대신에 소량의 uracil을 함유한 DNA가 합성되게 된다.
- 이와 같은 dut-, ung- strain에서 만든 single-stranded phagemid DNA를 template로 하고 mutation을 함유한 oligonucleotide를 primer로 시험관내에서 double-stranded circular DNA를 합성하여 wild type E.coli에 넣어주게 되면, 두 가닥의 strand 중에 uracil을 함유한 phagemid DNA는 선택적으로 제거되고 시험관에서 합성된 DNA가 template로 이용되어 복제가 일어난다.
- 이 방법은 비교적 비용이 적게 든다는 장점이 있지만, uracil이 함유된 ssDNA를 제조하여야 한다는 불편함이 있다.
세번째 : PCR을 이용하여 mutation을 함유한 DNA fragment를 만들어 subclone을 제조하는 방법
- 네개의 primer를 합성해야 하므로 비교적 비용이 많이 드나 요즘 oligonucleotide 합성 가격이 내려가서 비용 부담이 적어졌다.
## 세가지 방법 모두 하루에 transformation까지 할 수 있고 높은 성공률을 보장하므로 어떤 방법을 이용하여도 안전하다.
'전공이야기 > 실험방법' 카테고리의 다른 글
| PCR-4primers 를 이용한 Site-directed mutagenesis 실험 방법 정리 (0) | 2023.01.17 |
|---|---|
| Cell cycle analysis [FACS - PI staining] 실험 방법 정리 (3) | 2023.01.17 |
| RT-PCR (Revers Transcription PCR) 실험 방법 정리 (0) | 2022.12.06 |
| RNA 정량 (RNA 농도 측정) 방법 및 요약 정리 (0) | 2022.12.06 |
| RNA purification (RNA 정제) 어렵지 않아요~! 실험 진행 시 주의 사항 핵심 정리 요약입니다. (0) | 2022.12.06 |




댓글